


Librería fundamental sobre la cual se construye todo el ecosistema científico y de análisis de datos en Python.
Numpy básicamente ofrece:
Python está organizado en módulos que son archivos con extensión .py que contienen funciones, variables y otros objetos.
Y en paquetes o bibliotecas, que son conjuntos de módulos. Cuando queremos utilizar objetos que están definidos en un módulo tenemos que importarlo.
import numpy as np
import matplotlib.pyplot as plt
%matplotlib inline
"""1. Definimos una función"""
def f(x):
return (x**3) * (np.cos(x))
"""2. Definimos un dominio X, esto genera un array de numpy,
en este caso un array unidimensional, es decir un vector"""
x = np.arange(-10, 10, 0.1)
# Para limpiar la salida de una celda, selecciónela, digite
# ESC y luego sin soltarlo, pulse la letra R y luego la Y
"""3. Evaluamos la función en el array x"""
y = f(x)
"""4. Mostramos la gráfica"""
plt.plot(x, y, label='$x^3 * cos(x)$')
[<matplotlib.lines.Line2D at 0x7f11706c4c40>]
"""5. Incluir más detalles en la gráfica"""
plt.plot(x, y, label='$x^3 * cos(x)$')
plt.title('Comportamiento de f(x)')
plt.xlabel('$Dominio$')
plt.ylabel('$Rango_5$')
plt.grid()
plt.legend()
plt.show()
N,M = 100, 100
a = np.random.rand(10000)
a = a.reshape(N, M) # reshape es un método nativo de los arrays de numpy
b = np.random.rand(10000).reshape(N, M)
c = np.empty([100,100])
%%timeit
for i in range(N):
for j in range(M):
c[i,j] = a[i,j] + b[i,j]
%%timeit
c = a + b
¡1000 veces más rápido! Se hace fundamental vectorizar las operaciones y aprovechar al máximo la velocidad de NumPy.
Estas funciones operan sobre números y sobre arrays.
np.e
np.pi
np.log(2)
np.sin(np.pi/4)
np.sqrt(2)
np.linalg.norm(a)
Un array de NumPy es una colección de N elementos, igual que una secuencia en Python (por ejemplo, una lista). Tiene las mismas propiedades que una secuencia y algunas más.
Para crear un array, la forma más directa es pasarle una secuencia a la función np.array
np.array([1, 2, 3, 4, 5])
Los arrays de NumPy son homogéneos, es decir, todos sus elementos son del mismo tipo. Si le pasamos a np.array una secuencia con objetos diferentes, promocionará todos al tipo con más información. Para acceder al tipo del array, podemos usar la función dtype
a = np.array([1, 2, 3.0, 4, 5])
a.dtype
NumPy intentará automáticamente construir un array con el tipo adecuado teniendo en cuenta los datos de entrada, aunque nosotros podemos forzarlo.
np.array([1, 2, 3, 4, 5], dtype=float)
np.array([1, 2, 3, 4, 5], dtype=complex)
También podemos convertir un array de un tipo a otro utilizando el método .astype
a
a = a.astype(int)
a
Una vez se ha creado un array es posible acceder a información sobre su estado.
# Forma del array
a.shape
# Total de bytes que ocupa este array
a.nbytes
# Número de dimensiones de un array
a.ndim
# Suma de elementos
a.sum()
# Mínimo, máximo y media
a.min(), a.max(), a.mean()
a = np.array([1, 2, 3])
a
b = np.array([[1,2,3,4],[5,6,7,8],[9,10,11,12]])
b
b.ndim
b.shape
np.arange(10)
np.arange(0, 1, 0.2)
np.linspace(0, 1, 10)
np.ones((3,3))
np.zeros((2,2))
np.eye(4, 4)
np.diag(np.array([1,2,3,4]))
np.random.rand(4) # Uniforme entre [0, 1]
np.random.normal() # Gaussian de Media 0 y Varianza 1
np.random.normal(loc=0.0, scale=1.0, size=[4,4])
np.random.randint?
np.random.randint(low=1, high=20, size=5)
# Note qué pasa si rodamos esta celda nuevamente
np.random.seed(1234)
np.random.randint(low=1, high=20, size=5)
lena = plt.imread('images/lena.png')
print(type(lena))
print(lena.shape)
lena
Una de las herramientas mas importantes a la hora de trabajar con arrays es el indexado. Consiste en seleccionar elementos aislados o secciones de un array. Nosotros vamos a ver la indexación básica, pero existen técnica de indexación avanzada que convierten los arrays en herramientas potentísimas.
a = np.arange(10)
a
a[0], a[2], a[-1]
a[::-1]
Para arrays multidimensionales, los índices son tuplas de enteros según las dimensiones del array.
a = np.arange(16).reshape((4,4))
a
En 2D la primera dimensión corresponde a las filas, la segunda a columnas
a[0, 0]
No solo podemos recuperar un elemento aislado, sino tambien porciones del array, utilizando la sintaxis [<inicio>:<final>:<salto>]
a[1:3:1, :]
a[:, 1:3:1]
plt.imshow(lena, cmap='gray')
<matplotlib.image.AxesImage at 0x7f11704b1e80>
plt.imshow(lena[128:384, 128:384], cmap='gray')
a = np.arange(5)
a
a + 1
b = np.ones(5)
a - b
Esto NO es multiplicación de matrices
c = np.ones(3)
c * c
(para multiplicación de matrices está la función np.malmut,
y para producto punto entre vectores np.dot)
a = np.array([1, 2, 3, 4, 5, 6, 7])
b = np.array([4, 2, 2, 4, 3, 2, 1])
a == b
a > b
Podemos usar estas comparaciones como una mascara para modificar y acceder a los valores que cumplan la condición.
a[a > b] = 0
a
a = np.arange(5)
a
np.sin(a)
np.exp(a)
a = np.array([1, 2, 3, 4])
np.sum(a)
a.sum()
a = np.arange(9).reshape((3,3))
a
# Primera dimensión
a.sum(axis=0)
# Segunda dimensión
a.sum(axis=1)
Funciona de igual manera con el axis y en múltiples dimensiones.
x = np.array([1, 2, 3, 4])
x.min()
x.max()
x.mean()
x.std()
tablero de ajedrez, con unos en las casillas negras y ceros en las blancas.Puede mostrar el tablero de ajedrez usando plt.imshow(array)
Las operaciones básicas (suma, resta, etc.) se hacen elemento por elemento y funciona en arrays de diferente tamaño.
La imagen siguiente da un ejemplo de broadcasting
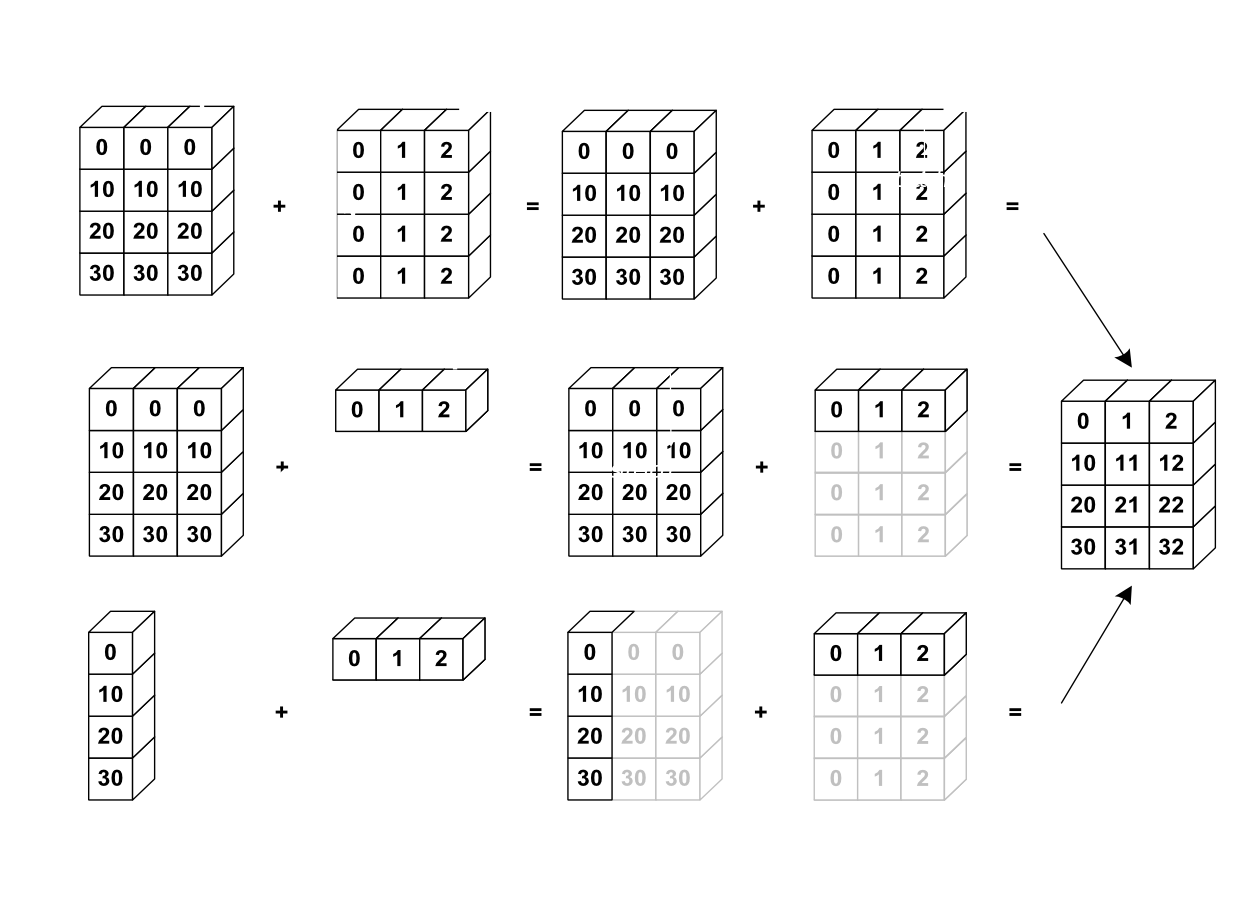
a = np.array([[0], [10], [20], [30]])
a
b = np.array([0, 1, 2])
b
a + b
a = np.array([[1, 2, 3], [4, 5, 6]])
a.ravel()
a.T
a.T.ravel()
La operación inversa a colapsar.
b = a.ravel()
b
b.reshape((2,3))
b.reshape((3,2))
# Leer datos desde archivo
data = np.loadtxt('data/populations.txt')
data
Genere un gráfico para observar el cambio en la población de liebres y de linces a lo largo de los años
# Almacenar datos en formato texto
# Suponga que se ha cometido un error, y los datos de población de linces,
# reportados en miles, se trataba en realidad de centenares. Corrija la
# columna respectiva y almacene la tabla con un nuevo nombre utilizando:
np.savetxt('data/pop2.txt', data, fmt='%5.i', delimiter='\t')
Numpy tiene su propio formato binario.
data = np.ones((3,3))
data
np.save('data/ones.npy', data)
data3 = np.load('data/ones.npy')
data3
array, arange, ones, zeros, randshape y los diferentes métodos de indexado para obtener diferente secciones del array array[::2], etc.reshape o aplanarla con ravela[a < 0] = 0array.max() o la media array.mean(), entre otras.Usando los resultados de un análisis químico de vinos obtenidos de la misma región en Italia pero de tres diferentes cultivos. Examine el archivo data/wine.csv.
Los atributos del dataset son:
Use np.set_printoptions(suppress=True, precision=3) para imprimer los datos de una manera mas legible.
data/wine.csv, use el parámetro delimiter=',' para valores separados por coma.3.5 se considera demasiado maduro, cuántos vinos tienen una concentración mayor?plt.hist(array) cree un histograma de ácido málico.plt.scatter(array1, array2) cree un gráfico de dispersión de las dos columnas normalizadas.np.set_printoptions(suppress=True, precision=3)
array([[ 1. , 14.23, 1.71, ..., 1.04, 3.92, 1065. ],
[ 1. , 13.2 , 1.78, ..., 1.05, 3.4 , 1050. ],
[ 1. , 13.16, 2.36, ..., 1.03, 3.17, 1185. ],
...,
[ 3. , 13.27, 4.28, ..., 0.59, 1.56, 835. ],
[ 3. , 13.17, 2.59, ..., 0.6 , 1.62, 840. ],
[ 3. , 14.13, 4.1 , ..., 0.61, 1.6 , 560. ]])